“碱基”华东师大科研团队填补碱基编辑领域最后一块拼图
今天,很高兴为大家分享来自媒体滚动的华东师大科研团队填补碱基编辑领域最后一块拼图,如果您对华东师大科研团队填补碱基编辑领域最后一块拼图感兴趣,请往下看。
华东师范大学生命科学学院李大力教授团队继2022年10月在国际著名学术期刊 Nature Chemical Biology 报道新一代精准安全的腺嘌呤碱基编辑器ABE9以及11月在国际著名学术期刊 Nature Biotechnology 报道高精度新型胞嘧啶碱基编辑系统Td-BEs后,一年不到又迎来新的技术突破,2023年6月15日该团队再次在 Nature Biotechnology 上发表了题为“Adenine transversion editors enable precise, efficient A·T-to-C·G base editing in mammalian cells and embryos”的研究论文。
该研究报道了一系列新型腺嘌呤颠换编辑工具(AXBEs和ACBEs),并且证明了ACBEs在不同细胞系与小鼠胚胎中的高效性与精确性,其中产生的小鼠疾病模型(A>C平均效率44%-56%)等位基因突变高达100%。AXBEs和ACBEs为多元化的遗传操作和人类第二大类单核苷酸变异(SNVs)的基因治疗提供新的工具。
 Nature Biotechnology 刊发华东师大生命科学学院李大力教授团队研究成果
Nature Biotechnology 刊发华东师大生命科学学院李大力教授团队研究成果人类的遗传疾病主要由基因突变造成,并且约58%为单碱基突变(SNVs)【1】。目前,不依赖DNA双链断裂和模板参与的单碱基编辑器(baseeditors)是治疗遗传病强有力的基因编辑工具。现有的碱基编辑技术CBEs(C-to-T),CGBEs(C-to-G), GBEs(C-to-G/C-to-A)和ABEs可实现胞嘧啶转换/颠换编辑以及腺嘌呤的转换突变,由于缺少切除肌苷的内源DNA糖苷酶,ABEs的产物纯度可达99%【2-5】。然而,仍有25%的人类单碱基突变遗传疾病需要精准的腺嘌呤颠换编辑(A-to-C或A-to-T)才能纠正,因此开发高效精准的腺嘌呤碱基编辑器具有重要意义。
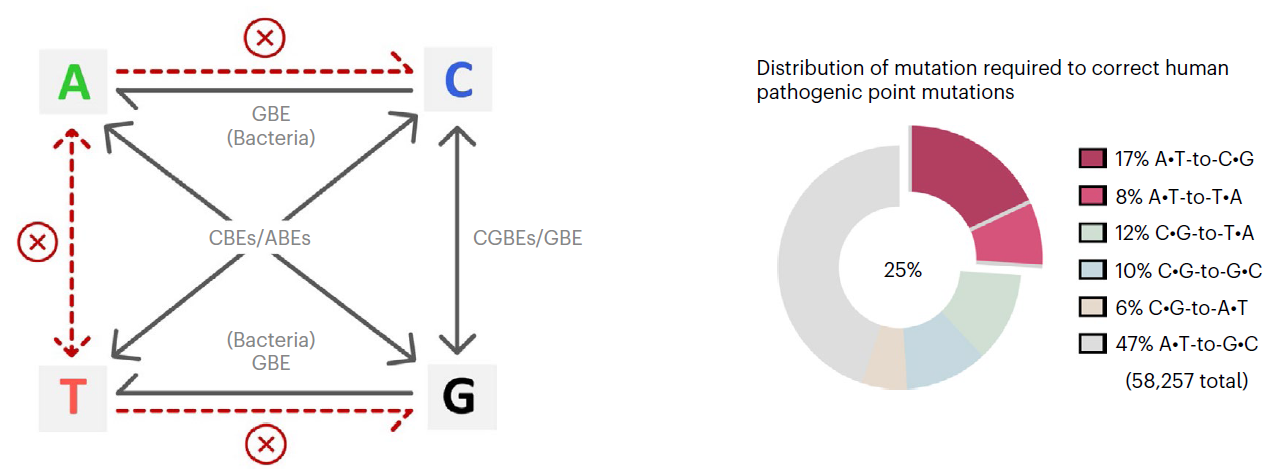 碱基编辑器介导碱基突变的种类及人类致病性点突变的分布
碱基编辑器介导碱基突变的种类及人类致病性点突变的分布碱基转换可通过碱基脱氨实现,而碱基颠换则需要依赖无嘌呤无嘧啶(AP)位点的创建,随后进行碱基切除修复途径(BER)而完成。鉴于内源糖苷酶低效的肌苷切除修复能力,华东师大研究人员希望寻找其他可将肌苷作为催化底物的酶,在大多数原核和真核生物中,DNA中的次黄嘌呤是由烷基腺嘌呤DNA糖基化酶或者核酸内切酶V去除,因此研究人员尝试将9种体外具有潜在肌苷切除活性的酶与腺苷脱氨酶TadA-8e和nCas9融合,结果惊喜的发现小鼠来源的烷基腺苷DNA糖苷酶(mAAG)可实现8.7%的A到Y(Y=C或T)的碱基颠换,并且大鼠、枯草芽孢杆菌来源的融合构建体也观察到了一定的腺嘌呤颠换突变。进一步的位置关系测试发现mAAG位于构建体C端的编辑活性最高,因此将其命名为AXBE(X代表任意碱基)。
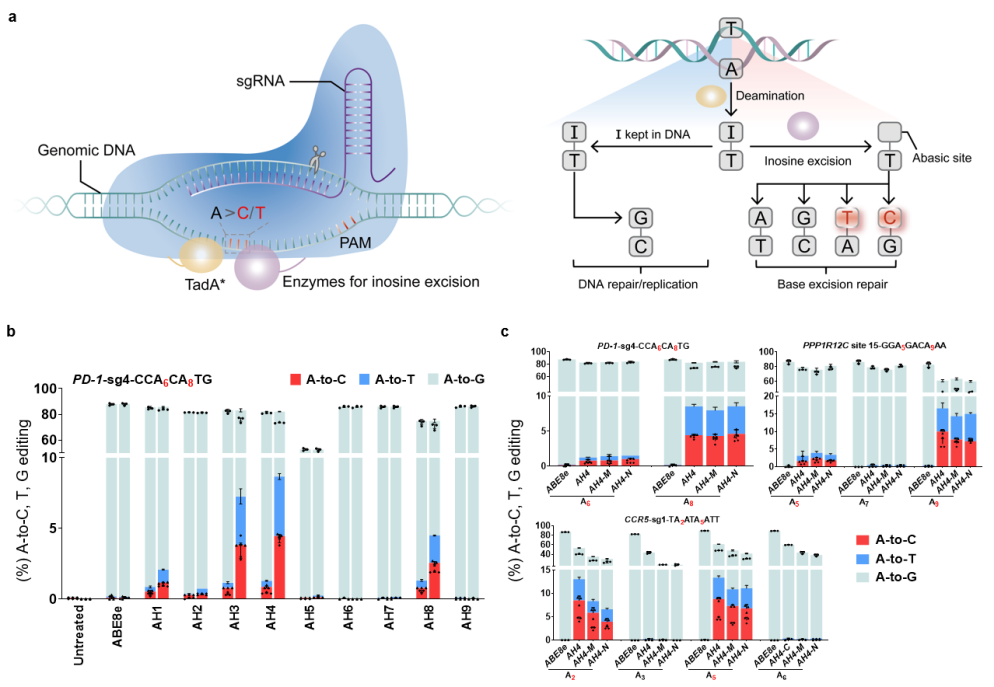 腺嘌呤碱基颠换原理图以及腺嘌呤颠换编辑器候选者筛选
腺嘌呤碱基颠换原理图以及腺嘌呤颠换编辑器候选者筛选经过大量内源性靶点评价以及体外酶活实验发现AXBE具有YA*R (R=A或G)碱基背景序列(motif)的偏好性。在含有YAR motif的靶点中,AXBE可产生15-32%的A到Y碱基颠换编辑。AXBE在HeLa细胞系中也可达到高达46%的腺嘌呤碱基颠换。通过Cas9依赖性/非依赖性DNA水平脱靶评估以及转录组水平脱靶检测发现,相比于ABE系统,AXBE的脱靶明显降低,尤其是在RNA水平上降低了90%。
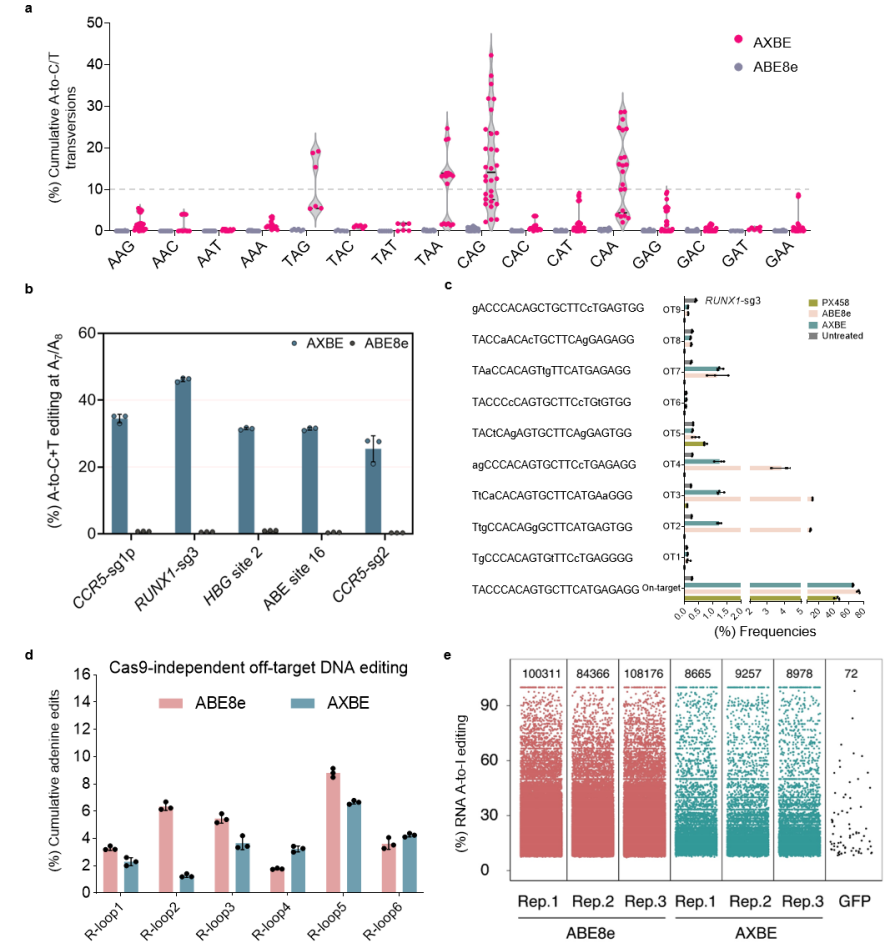 AXBE编辑特性的表征
AXBE编辑特性的表征为增加腺嘌呤颠换编辑效率并拓展靶向范围,基于结构导向的理性设计和筛选鉴定出mAAG中的两个关键突变(mAAG-EF)极大提高对其底物肌苷切除的活性,基于此产生的AXBEv2介导更高效的A到Y编辑,甚至在非YAR-motif位点的颠换突变也显著提升,因此有效改善了颠换编辑的序列背景选择性。此外,mAAG-EF也可以兼容PAM识别更广的其他Cas9变体(如spNG, spRY),表明了可以广泛地靶向基因组,扩大应用范围。
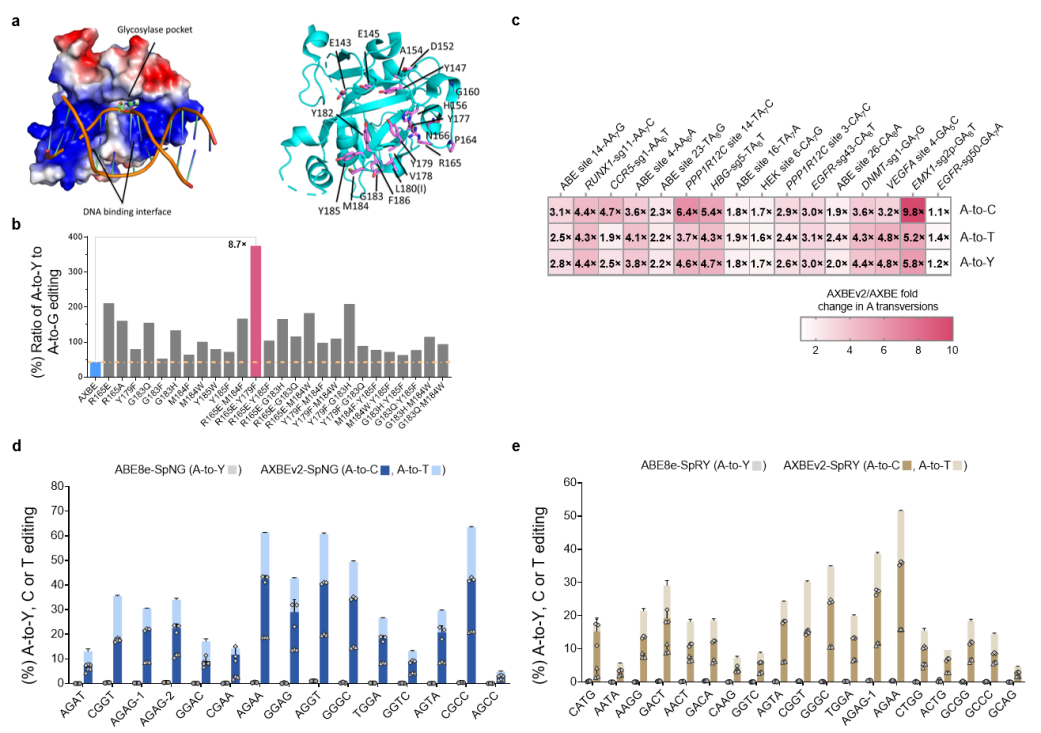 mAAG的分子进化以及AXBEv2编辑特性的表征
mAAG的分子进化以及AXBEv2编辑特性的表征虽然AXBEv2展现了高效的A到Y编辑能力(C为主要产物),但也诱导了严重的非目标碱基A到G突变,于是研究人员希望通过缩窄编辑窗口减少不想要的编辑副产物。腺嘌呤脱氨酶的改造及Cas9嵌入策略的尝试使得A到G的编辑副产物大幅降低,并且A到C的编辑效率还进一步提高,因此该类型编辑器被命名为ACBEs。其中ACBE版本最高可实现45%的A到C编辑以及73%的碱基颠换编辑,而ACBE-Q版本(TadA-8e中引入N108Q突变)相较于ACBE,进一步显著减少了旁观者A到G突变,更加精准地编辑sgRNA的A4-A6位,精确度最高提高了171倍,且只产生背景水平的Cas9非依赖性脱靶事件(平均脱靶效率<0.3%),展现出较高的应用安全性。
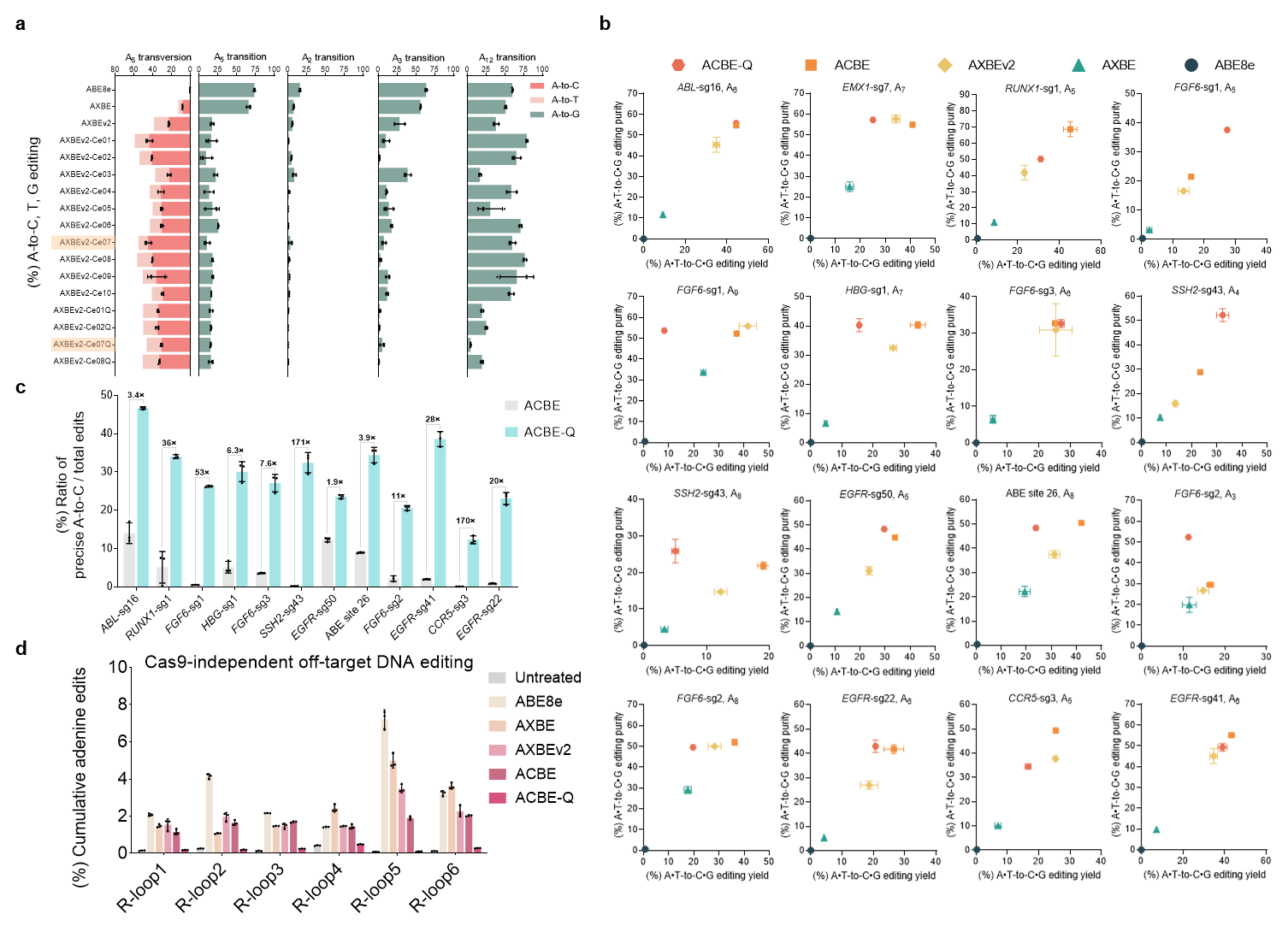 ACBEs编辑器的开发及表征
ACBEs编辑器的开发及表征此外,ACBE-Q在小鼠体内也展现出极高的编辑效率和精度。在构建杜氏肌营养不良小鼠疾病模型中,70%突变小鼠(21/30只)实现了靶位点A6到C的编辑,平均编辑效率为56%,A>C编辑纯度最高可达到99.8%。更重要的是,传统的终止密码子产生只能依赖于胞嘧啶的突变,该研究证明了ACBEs可在富含AT的序列背景中基于腺嘌呤引入提前成熟的终止子,通过终止Tyr基因表达成功构建了小鼠白化病模型。最后研究人员评估了ACBEs编辑人类致病性SNVs位点的能力,结果表明ACBE在SH3TC2基因中有效产生63%的终止密码子,该基因的终止会导致腓骨肌萎缩症疾病,通过靶向MYO7A基因,ACBE精准产生与耳聋有关的L618R错义突变。为了研究ACBEs的治疗潜力,研究者构建了携带STAT3c.1145G>T(该热点突变引起复发性传染病)突变的稳转细胞系,ACBEs可以在目标位置引入了想要的A到C纠正编辑。
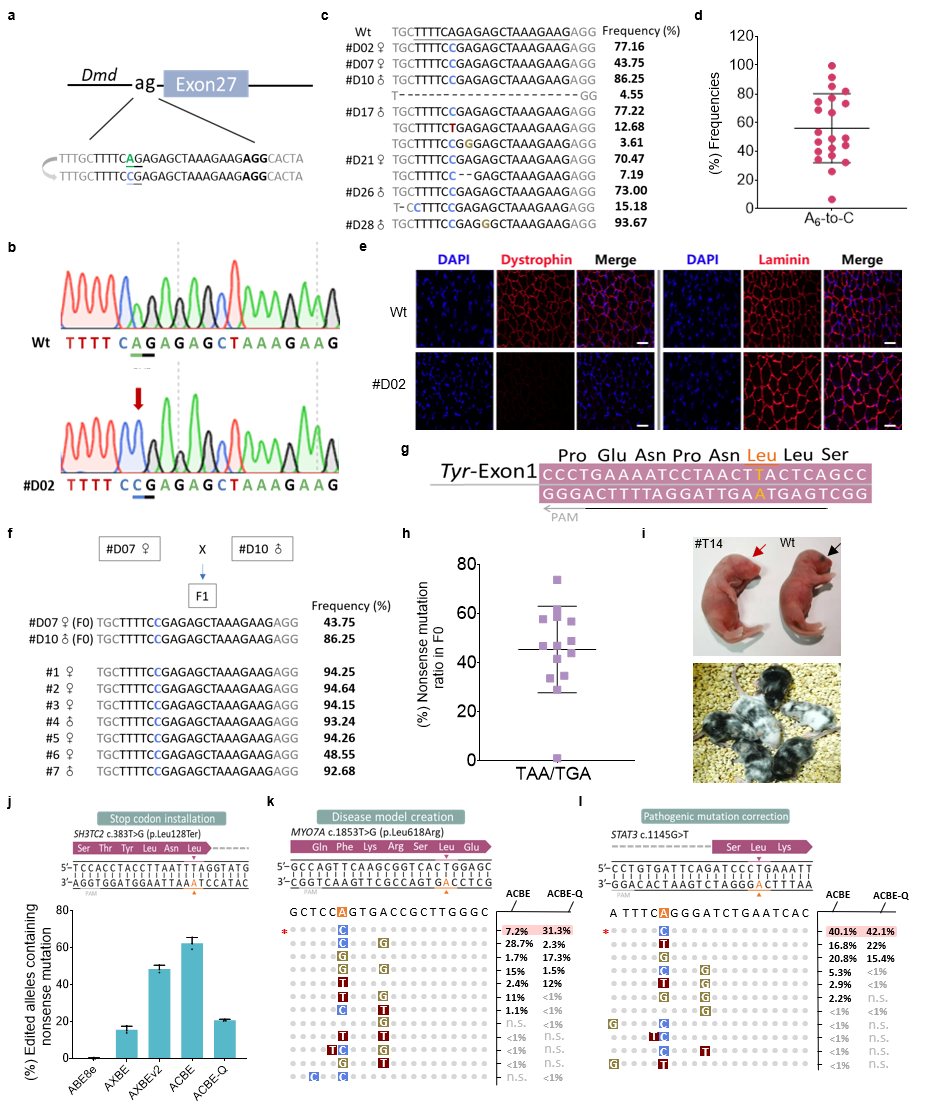 ACBEs在小鼠体内精确编辑以及在细胞系中模拟或纠正人类致病SNVs
ACBEs在小鼠体内精确编辑以及在细胞系中模拟或纠正人类致病SNVs总的来说,该研究通过筛选发现融合小鼠来源的烷基腺苷DNA糖苷酶(mAAG)的腺嘌呤颠换编辑器AXBE可在特定序列中实现腺嘌呤的颠换,进一步的分子进化使得A到Y编辑最高可达73%。AXBEs带来丰富的密码子和氨基酸改变,未来将更适用于分子进化、遗传筛选、谱系示踪等应用。ACBEs能够在AT富集区域提前引入终止密码子扩大了基因调控的范围,并且mAAG与不同Cas变体的兼容性有望进一步扩大A到C的靶向范围,表明了纠正人类第二大类致病性SNVs的治疗前景,另外ACBEs还可以作为独特的工具来探索与胸腺嘧啶相对的特定AP位点如何修复的机制。
华东师范大学生命科学学院博士毕业生陈亮、博士研究生洪梦佳和栾昌明为本论文的共同第一作者,华东师范大学为第一单位,华东师范大学生命科学学院李大力教授为本文通讯作者。麻省理工学院-哈佛大学博德研究所David R. Liu教授团队,华东师范大学生命科学学院刘明耀教授及宋高洁研究员、香港中文大学冯波教授等对本项研究提供了重要支持。该研究受到了科技部重点研发计划、国家自然科学基金以及上海市教委前沿科学基地和重大项目等支持。
附:
论文链接:https://www.nature.com/articles/s41587-023-01821-9
参考文献:
[1] Landrum, M.J. et al. ClinVar: public archive of interpretations of clinically relevant variants.Nucleic Acids Res. 2016, 44: D862-868.
[2] Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage.Nature, 2016, 533: 420-424
[3] Koblan LW, Arbab M, Shen MW, et al. Efficient C·G-to-G·C base editors developed using crispri screens, target-library analysis, and machine learning.Nat Biotechnol, 2021, 39: 1414-1425
[4] Zhao D, Li J, Li S, et al. Glycosylase base editors enable c-to-a and c-to-g base changes.Nat Biotechnol, 2021, 39: 35-40
[5] Gaudelli NM, Komor AC, Rees HA, et al. Programmable base editing of A·T to G·C in genomic DNA without DNA cleavage.Nature, 2017, 551:464-471.
好了,关于华东师大科研团队填补碱基编辑领域最后一块拼图就讲到这。
版权及免责声明:凡本网所属版权作品,转载时须获得授权并注明来源“科技金融网”,违者本网将保留追究其相关法律责任的权力。凡转载文章,不代表本网观点和立场,如有侵权,请联系我们删除。
相关文章
- “西湖”西湖大学成立5周年,施一公:做创新的守护者是使命也是未来
- “儿子”妈妈将50余万“读书钱”存儿子名下,19岁儿子取出转给女友,妈妈无奈起诉追回
- “力学”王博已任大连理工大学副校长
- “说了”云朵的话语,心灵的方剂 ——读周实《有些话语好像云朵》
- “超新星”云南天文台发现Ia型超新星前身星候选体
- “科幻”和成都和教育,一起遇见未来!两份重要“科幻教育”名单公布
- “红星”梁静茹南京演唱会再现“柱子票” 主办方:临时布置追光灯,正与观众沟通
- “导盲犬”视障女子自曝带导盲犬进公园遭保安阻拦 公园方:天色较晚不知其实情,沟通后已放行
- “小行星”首次!我国计划实施近地小行星防御任务
- “肿瘤”科学家揭示肿瘤免疫逃逸新机制,鉴定三个癌症生存相关因子,为肿瘤免疫治疗注入新动力
- “标准版”极空间Z4 Pro四盘位NAS大升级:酷睿N305处理器加持 性能提升至2.5倍
- “责任编辑”AI太火 NVIDIA躺赢!下一代GPU提前半年问世
- “责任编辑”腾势D9第10万辆车主公布:曹德旺之子曹晖
- “器官”转基因猪肾移植的猴子活了两年
- “空心菜”空心菜是抽筋菜吃完会抽筋?学者:谣言 仅一种人极低概率出现
- “疫苗”科普:为mRNA疫苗快速研发奠定基础——2023年诺贝尔生理学或医学奖成果解读
- “疫苗”为新冠疫苗开发贡献“加速度”!2023年诺贝尔生理学或医学奖解读
- “责任编辑”苹果扎心!用户反馈iPhone 15 Pro发热严重:不带壳没法用 原因众说纷纭
- “游戏”9月版号名单公布,中手游、心动等在列,Soul母公司取得首枚网游版号
- “责任编辑”3.6万元!雷蛇发布兰博基尼联名游戏本:全球限量150台









